De rode panda en Cserhati (15): de data en resultaten in het CRSQ artikel
In zijn Creation Research Society Quarterly artikel gebruikt Cserhati drie typen gegevens om de plaats van rode panda tussen andere soorten te vinden: Whole Genome K-mer Signature, mitochondriaal DNA sequentie, en aminozuurvolgorde in het eiwit cytochroom-b.
De eerste
twee sets gegevens, WGKS en mtDNA, zijn dezelfde als in zijn BMC Genomics
artikel. Voor de cytochroom-b aminozuurvolgorde gebruikt Cserhati nieuwe gegevens.
Er zijn dan gegevens over de volgende aantallen soorten/ondersoorten:
|
familie |
WGKS |
mtDNA |
aa
cytochroom-b |
|
katten Felidae |
11 |
- |
25 |
|
beren Ursidae |
5 |
15 |
5 |
|
marters Mustelidae |
10 |
30 |
17 |
|
wasberen Procyonidae |
0 |
2 |
1 |
|
stinkdieren Mephitidae |
1 |
3 |
1 |
|
rode panda Ailuridae |
1 |
2 |
2 |
|
|
28 |
52 |
51 |
De wasbeerfamilie ontbreekt bij de WGKS data, al had Cserhati voor twee soorten gegevens kunnen verzamelen. Het artikel van Tsuchiya et al in Genome Biology and Evolution met de genomen van de kinkajoe en de wasbeer is gepubliceerd in januari 2021. Dit lijkt op tijd voor bewerking voor een artikel in het najaar van 2021
WGKS
Uit de WGKS gegevens berekent Cserhati een correlatiematrix tussen de soorten. Dit is dezelfde correlatiematrix als in het BMC Genomics artikel, zoals blijkt uit de twee Additional Files. Cserhati geeft heatmaps van de correlatie matrix in beide artikelen: dezelfde figuur, maar anders ingekleurd.
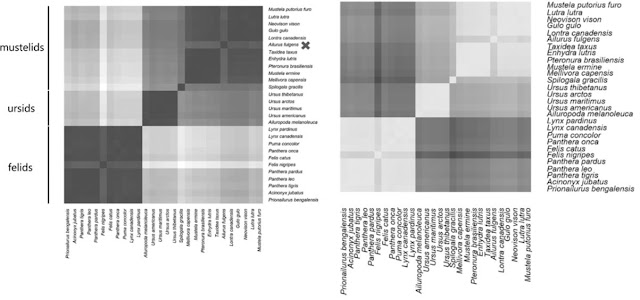 |
| Figuur 1. Heatmaps van de correlaties op de WGKS data: links als in CRSQ artikel, rechts als in BMC Genomics artikel. De grijsschaal is omgedraaid, verder is het dezelfde figuur. |
De plaats van de rode panda Ailurus fulgens staat rechts aangegeven, midden tussen de marterachtigen. Dit leidt tot een soort 'Deense vlag' patroon. De enige soort van de stinkdierfamilie Mephitidae in deze data, Spilogale gracilis, is te vinden tussen de beren en de marters, het blokje naast de 'Deense vlag'.
In deze figuur geeft Cserhati families
aan: felids, de familie katten Felidae, ursids, de familie beren Ursidae, en
mustelids, de familie marterachtigen Mustelidae. Kennelijk worden in de figuur
de rode panda en Spilogale tot de
marterfamilie gerekend.
In de tekst zegt Cserhati dat er drie
clusters gevonden worden in de WGKS data: de katten, de beren en de superfamiie
Musteloidea:
...
felids covering the family Felidae (eleven species), ursids covering the family
Ursidae (five species), and musteloids, a superfamily including Mephitidae and
Mustelidae (twelve species).
Er
zijn twee opmerkelijke punten bij Cserhati's resultaat 'drie clusters'.
Het eerste opmerkelijke punt is dat Cserhati hier in het CRSQ artikel tot drie clusters komt, en in het BMC Genomics artikel op precies dezelfde resultaten vier clusters geeft. In het BMC Genomics artikel wordt het stinkdier Spilogale gracilis apart gezet als vierde cluster. Hier wordt Spilogale gracillis bij het grote cluster Musteloidea gerekend. Cserhati geeft silhouette plots voor 3 clusters en voor 4 clusters: vier met Spilogale als afzonderlijk cluster. De silhouette-wijdte nul voor Spilogale geeft aan dat clustering in het naastliggende cluster beter is. De silhouette plots laten betere clustering met drie clusters zien.
Het tweede opmerkelijke punt is dat Cserhati de rode panda indeelt bij de marterfamilie Mustelidae - iets dat hij had moeten aantonen, maar hier zonder argumentatie aanneemt. De rode panda verschilt van alle soorten marterachtigen: het is duidelijk te zien in de heatmap.
Uit figuur 2 en 4 blijkt duidelijk dat de grote verschillen zijn tussen de katten linksonder en alle andere soorten. Om verschillen binnen de superfamilie Musteloidea helder te krijgen, is het beter om op dezelfde correlaties een heatmap voor de Musteloidea allen te maken.
Het verschil is statistisch significant (blogpost Cserhati 9). De rode panda mag dan clusteren binnen de superfamilie Musteloidea, het is geen mustelid, geen marterachtige.
mtDNA base sequenties
Ook
uit de mitochondriaal DNA sequentie gegevens berekent Cserhati een
correlatiematrix tussen de soorten. Dit is dezelfde correlatiematrix als in het
BMC Genomics artikel. Cserhati geeft een heatmap van de correlatiematrix: anders
ingekleurd, met de soorten anders gerangschikt.
Cserhati geeft de volgende interpretatie:
In this
heatmap we can see two main, large clusters as well as three smaller ones.
Met de twee
grote clusters zullen de beren ursids en de marterachtigen mustelids zijn bedoeld.
De interpretatie met twee grote clusters en drie kleine clusters wordt
aannemelijk gemaakt door de plaatsing van de twee rode panda ondersoorten
linksonder (Ailurus) en de plaatsing
van de twee wasbeerachtigen (procyonids) en de drie stinkdieren (mephitids)
rechtsboven. Zo is moeilijk te zien dat de correlaties van de rode panda met de
mustelids, mephitids en procyonids lichtgrijs kleuren langs de onderrand,
donkerder dan bij de beren.
De
correlaties zijn te vinden in Additonal File 2 van het artikel. Ik heb de
volgorde van de families langs de assen veranderd, en de correlatie matrix wat
helderder ingekleurd voor een duidelijker heatmap.
Nu hebben we een helder plaatje. Er zijn twee grote groepen, met de berenfamilie Ursidae linksboven en rechtsonder de superfamilie Musteloidea met de families Mephitidae stinkdieren, Ailuridae rode panda, en Procyonidae wasberen. Binnen de Musteloidea lijkt meer variatie te bestaan dan binnen de beren, namelijk de families en onderfamilies.
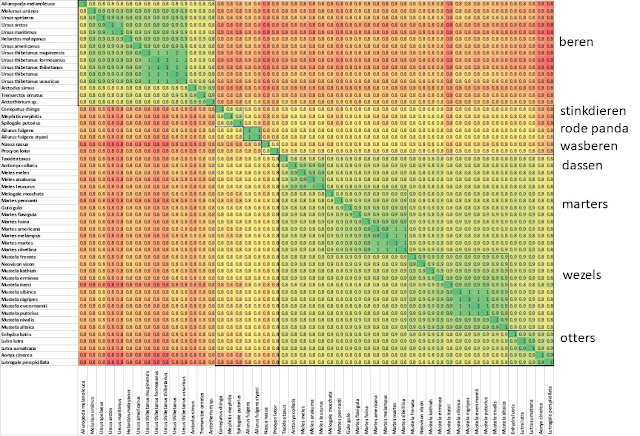 |
| Figuur 9. Heatmap uit figuur 8 met families en onderfamilies binnen de Musteloidea aangegeven. |
Er zijn wat verschillen zichtbaar tussen de familie marterachtigen Mustelidae en de andere drie families in de superfamilie Musteloidea. Het is iets meer oranje in die banen dan in het vierkant. Dat kan uitvergroot.
Het is
mogelijk een heatmap te maken van de superfamilie Musteloidea op zichzelf. De
grote verschillen met de beren zijn dan uit de kleuring van de heatmap
verdwenen. De verschillen binnen de Musteloidea worden zichtbaar bij voortgezette
heatmap kleuring.
 |
| Figuur 10 Dezelfde correlatiematrix als in de figuren 7, 8 en 9 met de verschillen in grootte van de correlaties binnen de Musteloidea aangegeven. |
De verschillen tussen de familie marterachtigen Mustelidae en de andere drie families in de superfamilie Musteloidea zijn nu sprekend. De vier onderfamilies binnen de familie marterachtigen zijn helder zichtbaar.
Dit kan nog
verder doorgevoerd, met een heatmap kleuring voor alleen de Mustelidae. Dan
komen verschillen in mtDNA tussen de onderfamilies nog wat beter uit.
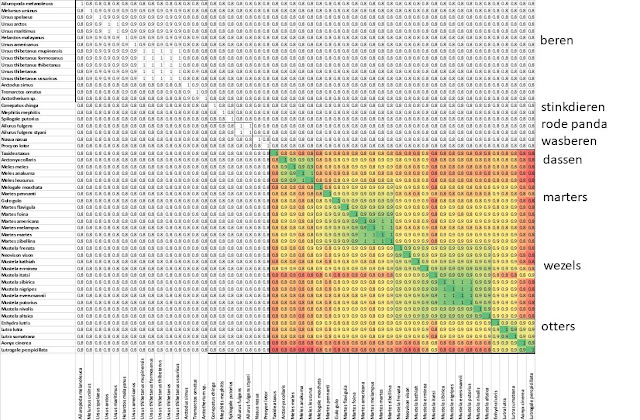 |
| Figuur 12 Dezelfde correlatiematrix als in de figuren 7, 8, 9 en 10 met de verschillen in grootte van de correlaties binnen de Mustelidae aangegeven. |
Zouden de otters toch ook wat apart staan? Tenslotte zijn otters 'biologically meaningful' verschillend van de andere soorten hier - zwemmers en viseters.
Cserhati interpreteert figuur 7 (zijn figuur 3) alsof de vijf bekende taxonomische families uit de clustering komen.
In this
heatmap we can see two main, large clusters as well as three smaller ones.
Er is geen
statistische onderbouwing van vijf clusters. Cserhati zegt vijf clusters, maar
dan verwacht je een silhouette plot dat dat duidelijk maakt. Uit de silhouette
plots die Cserhati geeft blijkt iets anders.
In het BMC
Genomics artikel staat in het mtDNA deel midden tussen de fylogenetische bomen de zin:
Supplementary
figure 3 shows the average silhouette width according to the number of
clusters, with an average silhouette width of 0.51 for two clusters.
Cserhati heeft silhouette plots voor verschillende aantallen clusters gemaakt, zoals te zien is in Supplementary figure 3 van het CRSQ artikel.
In feite vindt Cserhati twee grote clusters, namelijk de berenfamilie Ursidae en de superfamilie Musteloidea. Zo geeft hij het ook aan in zijn tabel met de statistische gegevens van de clusters, Table 3 van het BMC Genomics artikel.
Vijf clusters, de vijf klassieke families Ursidae beren, Mephitidae stinkdieren, Ailuridae rode panda, Procyonidae wasberen, Mustelidae marterachtigen is niet beter dan acht clusters. Acht clusters met de vier families Ursidae beren, Mephitidae stinkdieren, Ailuridae rode panda, Procyonidae wasberen en vier onderfamilies, Melinae dassen, Guloninae marters, Lutrinae otters en Mustelinae wezels binnen de marterfamilie is ook mogelijk, even goed als vijf clusters.
Aminozuurvolgorde in het eiwit
cytochroom b.
Het
mitochondriaal DNA bevat onder andere het gen voor het eiwit cytochrome b.
Cserhati besluit de aminozuurvolgorde van dit eiwit voor clustering van soorten
te gebruiken.
Cytochrome-b
is a structurally conservative protein which does not mutate freely. It is
often used to infer phylogenetic relationships between organisms (Meyer, 1994),
and can so be used in baraminology studies as well.
Cytochroom b
was een van de eerste genen waarvoor goede technieken en goede gegevens
beschikbaar kwamen. We hebben het dan wel over populariteit 30 jaar geleden,
voordat DNA sequenties van het hele mitochondriaal DNA beschikbaar kwamen.
Genbank en de NCBI database hebben duizenden entries; data vinden is niet
moeilijk.
Cserhati vergelijkt de aminozuurvolgorde in 25 katten, 5 beren (maar niet de reuzenpanda), de twee ondersoorten van de rode panda, het gestreept stinkdier Mephitis mephitis als enige van zijn familie, de neusbeer Nasua nasua als vertegenwoordiger van de wasbeerfamilie, en 17 soorten marterachtigen. De heatmap die Cserhati geeft van de correlatiematrix voor deze soorten is de volgende figuur.
In the heatmap
(Figure 4) we can see three main clusters and two species by themselves, N. nasua and M. mephitis. In the lower left there is a tight cluster of 25
felids. In the upper right there is a cluster of five ursid species, and
another with 19 mustelid species.
Er worden
geen silhouette plots gegeven om de drie clusters katten, beren en
marterachtigen + panda's en twee niet geclusterde soorten te
onderbouwen. Een silhouette plot had duidelijk moeten maken waarom er geen
cluster Musteloidea gerapporteerd wordt. Gezien de heatmap is een cluster
Musteloidea een mogelijkheid.
De rode panda
wordt zonder meer tot de marterachtigen Mustelidae gerekend. Zoals blijkt uit
de heatmap (fig 15 hier, fig 4 Cserhati) is dit niet vanzelfsprekend. De twee ondersoorten
van de rode panda vormen duidelijk een blokje naaste de soorten van de familie
Mustelidae; ook de correlaties van de rode panda met de marterachtigen zijn
weer lager dan de correlaties van de marterachtigen onderling, zoals aan de grijswaarden
te zien is.
Dit wordt duidelijker in een rood-groen heatmap. De correlaties zijn te vinden in Additonal File 3 van het artikel. Ik heb de volgorde van de families langs de assen veranderd, en de correlatie matrix wat helderder ingekleurd voor een duidelijker heatmap.
Er is een homogene groep katten, de weinige beren zijn ook homogeen, en de superfamilie Musteloidea marterachtigen, de neusbeer, het stinkdier en de rode panda's blijkt nogal heterogeen. De neusbeer is de bovenste in de Musteloidea, dan volgt het stinkdier, en dan de twee ondersoorten rode panda's. Neusbeer en stinkdier verschillen van rode panda en marterachtigen, maar meer van de beren en de katten.
Alleen de superfamilie Musteloidea inkleuren kan ook.
 |
| Figuur 17. Als figuur 16 met alleen de Musteloida ingekleurd als heatmap rood-groen. |
En inkleuren van alleen rode panda en de familie marterachtigen geeft:
 |
| Figuur 18. Als figuur 16 met alleen de rode panda en de marterachtige ingekleurd als heatmap rood-groen. |
Het is niet zozeer dat de rode panda bij de familie marterachtigen hoort, als wel dat de neusbeer Nasua nasua zoveel verschilt van de andere soorten uit dezelfde superfamilie.
Cserhati
gebruikt een vreemde selectie soorten om na te gaan of de rode panda aan de
hand van cytochroom b bij de marterachtigen zou horen. Waarom 25 soorten
katten? Die katten doen er niet veel toe in de analyse; hoogstens drijven ze de
niet-katten in een heatmap naar kleuren die dichter op elkaar zitten. Als we
willen weten waar de rode panda binnen de superfamilie Musteloidea geplaatst
moet worden, moeten we soorten uit de Musteloidea gebruiken.
Cserhati geeft zijn Fasta file met aminozuurvolgordes voor cytochrome b niet,wel de nummers van de accessies die hij gebruikt heeft. Ik heb de aminozuur volgorde van cytochroom b voor de marterachtigen, beren, rode panda, stinkdier, neusbeer en zeven soorten katten opgezocht in de NCBI database. Dit heb ik aangevuld met de aminozuurvolgorde van een ander stinkdier, en tien soorten van de wasbeerfamilie Procyonidae. Dit geeft de volgende heatmap:
De superfamilie Musteloidea blijkt ook hier behoorlijk heterogeen. In meer detail:
Binnen de familie Procyonidae komen sommige soorten vrij veel overeen met de famiie Mustelidae, maar verschillen andere soorten sterk van de Mustelidae. Een van de soorten die sterk verschillen is de neusbeer Nasua nasua. De keuze voor alleen de neusbeer in de heatmap van Cserhati doet de rode panda meer op de marterachtigen lijken dan over het geheel van de Procyonidae gerechtvaardigd is.
Conclusies
De WGKS
clustering geeft drie groepen: de familie katten Felidae, de familie beren
Ursidae en de superfamilie Musteloidea.
De mtDNA
clustering geeft twee groepen: de familie beren en de superfamilie Musteloidea.
De cytochrome
b clustering geeft een cluster katten, een cluster beren en is onduidelijk over
de Musteloidea.
De heatmaps
geven aan dat de rode panda in geen van deze drie analyses binnen de familie
Mustelidae valt.
Ook is te
zien dat clustering de biologie niet goed
weergeeft. De mtDNA analyse geeft twee clusters, maar de heatmap laat
duidelijk zien hoeveel detail er bij clustering verloren gaat.
Slordigheden:
Blz 78: whole genome sequence voor
Procyonidae is niet gedownload
Blz 79, Table 1: Spilogale gracilis bij cluster 3, de katten
Blz
81: "... the WGKS analysis, where the average silhouette width was 0.8 for
classifying M. mephitis ..." Het is Spilogale gracilis in de WGKS analyse, niet Mephitis mephitis.
In
figuur 2 is de tekst is hier niet in overeenstemming met de figuur. De
figuur had links 'musteloids', niet 'mustelids' moeten geven.
***
Cserhati, M.,
2021, Classification of the Enigmatic Red Panda (Ailurus fulgens) Based on
Molecular Baraminology-Based Analysis, Creation Research Society Quarterly 58
(2): 76-84
M.T.N Tsuchiya, R.B Dikow, K.P Koepfli, P.B Frandsen,
L.L Rockwood, J.E Maldonado, 2021. Whole-Genome Sequencing of Procyonids
Reveals Distinct Demographic Histories in Kinkajou (Potos flavus) and Northern
Raccoon (Procyon lotor) Genome Biology and Evolution, Volume 13, Issue 1,
January 2021, evaa255, https://doi-org.proxy.library.uu.nl/10.1093/gbe/evaa255.
Published: 17 December 2020
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-10-wasberen.html
https://en.wikipedia.org/wiki/Silhouette_(clustering)
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-9-clusters-op.html
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-11-clustering.html
Hennigan, T.
2010. The case for holobaraminic status in bears (family Ursidae) and the
implications within a creation model of ecology. CRSQ 46(4):271–283.
Meyer, A.
1994. Shortcomings of the cytochrome b gene as a molecular marker. Trends in
Ecology & Evolution 9(8):278–280














Reacties
Een reactie posten