De rode panda en Cserhati (12): fylogenetische bomen op grond van mtDNA
Cserhati presenteert drie 'hierarchical trees' op grond van mitochondriaal DNA. Hij gebruikt het volledige mtDNA zoals opgegeven in GenBank voor 52 soorten: 15 soorten en ondersoorten van de familie beren, de twee ondersoorten van de rode panda, drie soorten stinkdieren, 30 soorten van de familie marterachtigen, en nu ook twee soorten van de familie van de wasbeer, de wasbeer zelf en de neusbeer.
Cserhati
gebruikt met drie verschillende methoden om zijn 'hierarchical trees' te
krijgen. (Cserhati noemt zijn resultaten geen fylogenetische boom of fylogenie,
zie zijn onderschriften bij zijn figuren).
Hiërarchische
bomen dus, met drie fylogenetische methoden: UPGMA, NJ en ML. Fylogenetische bomen.
UPGMA en NJ
gebruiken de verschillen, afstanden, tussen sequenties.
UPGMA is de
simpelste en oudste methode om een fylogenetische boom uit sequentie data te
construreren. Het grote nadeel van UPGMA is dat deze methode veronderstelt dat
de veranderingssnelheid van de sequenties gelijk is in de tijd en over alle
lijnen, dus over de gehele fylogenetische boom. UPGMA produceert een impliciet
gewortelde boom.
In zijn
figuur 4 geeft Cserhati een UPGMA fylogenetische boom
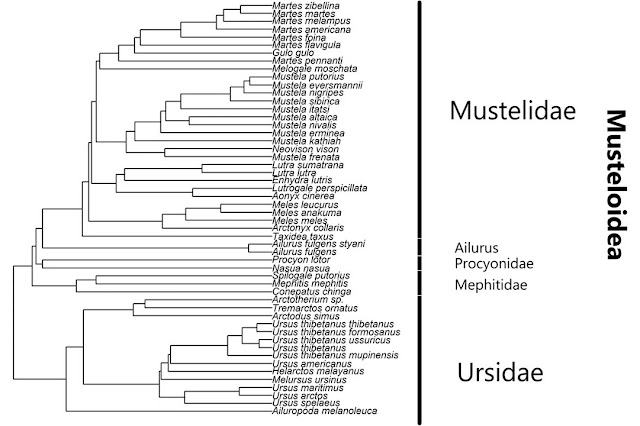
Figuur 1: Figuur 4 uit Cserhati BMC Genomics. UPGMA fylogenetische boom.
Figuur 2: figuur 4 van Cserhati BMC Genomics met opeenvolgende splitsingen binnen de Musteloidea.
Het bijschrift van Cserhati is:
UPGMA-based
hierarchical tree for the 52 species analyzed in the mtDNA study, based on
sequence identity metrics. Mustelids and ursids form two large clades, and
mephitids, procyonids forming two small groups. Ailurus fulgens and Ailurus fulgens
styani appear either to form their own clade, or loosely associate with
mustelids.
In de figuur
staat dat de familie Ailuridae, met de twee ondersoorten van de rode panda
Ailurus fulgens en Ailurus fulgens styani, de zustergroep is van de familie
Mustelidae. Dus hun eigen clade, hier als zustergroep van de marterachtigen. Dit
is de enige keer dat de Ailuridae als zustergroep van de marterachtigen
verschijnen in aanwezigheid van de Procyonidae, wasbeerfamilie.
Neighbour-Joining
neemt de afstanden tussen sequenties, paart de twee sequenties met de kleinste
afstand,en gaat verder met het paar alsof het een eenheid is. NJ turft steeds
waar de kleinste afstand te vinden is. De NJ methode geeft ongewortelde bomen,
maar er is geen veronderstelling over veranderingssnelheid in de tijd.
Een NJ programma
plot een ongewortelde boom als een cladogram: het programma voert een fictieve
wortel in. Het is aan de bewerker van de data om te zorgen dat de boom wortelt.
Cserhati schrijft dat hij de default instellingen gebruikt. Hij heeft dan een
ongewortelde boom geplot als een cladogram: de lengtes van de horizontale
lijnen zijn dan geen weergave van aantal verschillen tussen de soorten. De
lay-out geeft dan geen uitslag over de fylogenie. Cserhati geeft aan hoe goed
de splitsingen onderbouwd zijn.
Cserhati's omschrijving van zijn resultaat laat zien dat hij geen inzicht heeft in lay out en fylogenetische bomen:
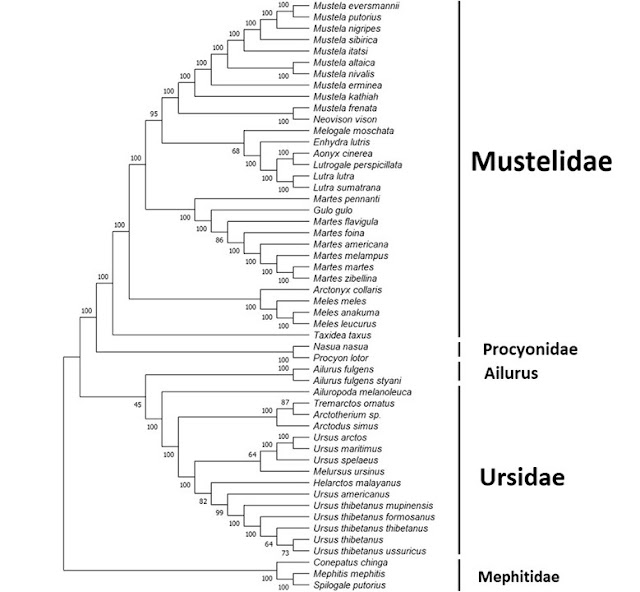
Mustelidae
forms a well-defined clade, with almost all branch points supported with a
bootstrap value of 100. Nasua nasua
and Procyon lotor form a smaller
clade right next to Mustelidae. The three mephitids, Conepatus chinga, Mephitis
mephitis and Spilogale putorius
also form a small clade, well separated from the other clades. The NJ method
places Ailurus next to Ursidae,
suggesting that they possibly form a monophyletic group. However, the node
connecting Ailurus with Ursidae only
has a bootstrap value of 45.
Een
fylogenetische boom is een mobile, en we kunnen om te beginnen de Mephitidae,
de stinkdieren, omklappen naar boven. Ook hebben we een ongewortelde boom, dus
we kunnen een wortel zetten tussen de beren Ursidae en alle andere soorten, die
allemaal tot de Musteloidea behoren. De lage bootstrap waarde van de knoop
tussen beren en Ailurus pleit hier
ook voor.
We nemen de
beren als buitengroep ten opzichte van de Musteloidea, en lopen langs de lijnen
om de opeenvolgende splitsingen in de Musteloidea te vinden. Dan komt een van
de bekende indelingen van de Musteloidea te voorschijn, nl die als in Flynn et
al (2000).
 |
| Figuur 4: Figuur 5 van Cserhati BMC Genomics met wortel tussen berenfamilie en Musteloidea superfamilie toegevoegd, en opeenvolgende splitsingen in de Musteloidea genummerd. |
In blogpost 2
staat een lijstje met artikelen die deze indeling van de Musteloidea vonden. De
uitkomst van de NJ fylogenetische boom van Cserhati hoort hier bij. Cserhati
laat zich misleiden door de lay-out die uit zijn programma komt.

Figuur 5. Schema van de indeling van de Musteloidea bij invoeren van de beren als buitengroep.
Maximum Likelyhood zoekt een fylogenetisch model dat het best past bij de gegevens. Dan is er een beginboom nodig:
Initial
tree(s) for the heuristic search were obtained automatically by applying
Neighbor-Join and BioNJ algorithms to a matrix of pairwise distances
Dat houdt in
dat Cserhati begon met een ongewortelde boom te maken - daarmee ook geen
wortel krijgt. Er is geen buitengroep in zijn ML fylogenetische boom.
Cserhati's omschrijving van zijn resultaat geeft nu de rode panda mogelijk een een eigen clade vormt:
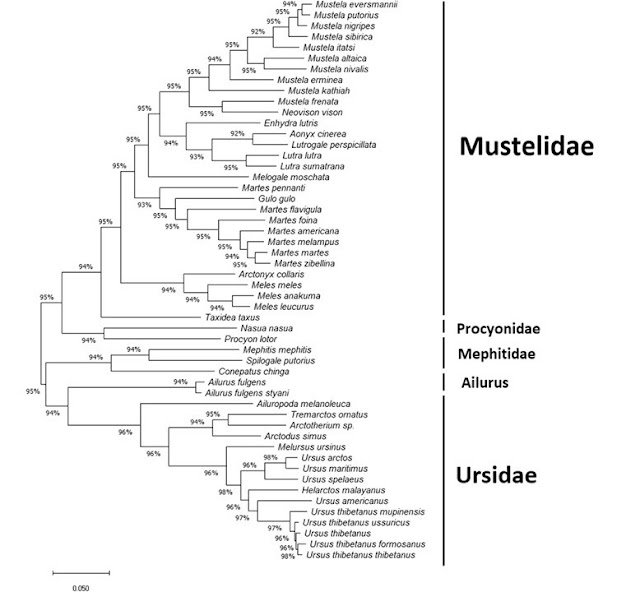
Here Mustelidae,
Procyonidae, and Mephitidae all form their own clades with a likelihood value
of at least 94%. As opposed to the NJ tree, here Ailurus is separated from
Ursidae suggesting that it might form its own clade as well.
De rode panda's krijgen een eigen clade met ook een likelihood
value van 94%, volgens de figuur.
De topologie
van de beren en de rode panda is hetzelfde in Cserhati's NJ boom als in
Cserhati's ML boom: het verschil is dat nu de genetische afstand ook zichtbaar
is. Cserhati's "opposed" en "separated" is gebaseerd op
onvoldoende inzicht in wat de fylogentische bomen geven.
Natuurlijk
kunnen we de beren weer als buitengroep ten opzichte van de Musteloidea
beschouwen, en daar de wortel van de fylogenetische boom plaatsen.
Het volgen van de lijnen van de ML fylogenetische boom om de opeenvolgende splitsingen in de Musteloidea te vinden levert dezelfde indeling als de NJ fylogenetische boom, namelijk die als in Flynn et al (2000). Zie figuur 5 hier voor het schema.
In totaal
vinden we voor de drie fylogenetische bomen op mtDNA van 15 beren en 37
musteloiden dat in alle drie bomen de rode panda een eigen groep, een clade,
vormt, namelijk de familie Ailuridae. De rode panda als afzonderlijke familie
is in 1996 voorgesteld door Ledje en Arnason; sindsdien is de status van de rode panda als familie Ailuridae geen onderwerp van
discussie (behalve voor Cserhati).
UPGMA geeft
een afwijkende fylogenetische boom, met de rode panda Ailuridae en niet de
wasbeerachtigen Procyonidae als zustergroep van marterachtigen Mustelidae. De
NJ fylogenetische boom en de ML fylogenetische boom geven de Procyonidae als
zustergroep van de Mustelidae, als algemeen gevonden wordt. De NJ en ML bomen
geven ook een indeling binnen de Mustelidae, in de plaatsing van otters,
wezels, dassen en marters, die overeenkomt met resultaten elders in de literatuur.
Cserhati geeft als conclusie:
The mtDNA
results as well as the maximum likelihood tree appear to place Ailurus fulgens into a monophyletic
group.
???
Misschien: de
clustering van de 52 soorten op hun mtDNA en alle drie fylogenetische analyses
van die 52 soorten op hun mtDNA geven de familie Ailuridae, de rode panda, als
zijn eigen monofyletische groep.
***
Cserhati, M., 2021, A tail of two pandas – whole
genome k-mer signature analysis of the red panda (Ailurus fulgens) and the
Giant panda (Ailuropoda melanoleuca), BMC Genomics 22: 228
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-021-07531-3
https://en.wikipedia.org/wiki/Computational_phylogenetics
https://en.wikipedia.org/wiki/Outgroup_(cladistics)
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-1-de.html
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-2-overzicht.htm
https://creationismeweersproken.blogspot.com/2023/01/de-rode-panda-en-cserhati-3-een.html
Ledje C, & Arnason U. (1996) Phylogenetic
relationships within caniform carnivores based on analyses of the mitochondrial
12S rRNA gene. Journal of Molecular Evolution 43:641–649.


Reacties
Een reactie posten